

Ein Künstliche Intelligenz-basiertes Klassifikationsergebnis eines radiologischen Bildes, etwa in Kategorien wie "krank" oder "gesund", ist typischerweise für den Nutzer bzw. die Nutzerin zunächst nicht nachvollziehbar. Die hierzu meist benutzten neuronale Netze agieren als "Black Box" – die gelernten Merkmale eines Bildes, die zur Entscheidung des Netzwerkes führen, sind für den Menschen nicht erkennbar.
VRVis forscht seit mehreren Jahren an erklärbaren, intuitiven und zuverlässigen KI-Lösungen, welche die Entscheidungen nachvollziehbar und vertrauenswürdig machen. Hierfür entwickeln wir Methoden, die die Ursachen für solche Entscheidungen eines neuronalen Netzes für den Nutzer bzw. die Nutzerin sichtbar machen. Diese Methoden geben nicht nur medizinischem Fachpersonal ein zusätzliches Feedback über den Hintergrund der Entscheidung, sondern helfen auch Entwicklerinnen und Entwicklern, die Qualität des trainierten Netzwerkes zu überprüfen.
Publikationen
D. Major , D. Lenis , M. Wimmer , A. Berg , T. Neubauer , K. Bühler (2023): On the importance of domain awareness in classifier interpretations in medical imaging. IEEE Transactions on Medical Imaging
Videos
Domain Aware Medical Image Classifier Interpretation by Counterfactual Impact Analysis
Medienberichte
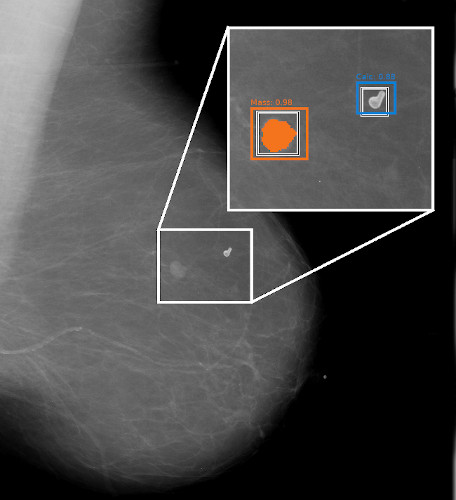
Brustkrebs ist weltweit die am häufigsten auftretende Krebsart bei Frauen. Daher ist die genaue Analyse von Mammographie-Bildern eine wichtige Aufgabe in der Radiologie, da besonders das frühzeitige Erkennen von Risikofaktoren und verdächtigen Veränderungen der Brust von großer Bedeutung für eine erfolgreiche Therapie sind.
In Kooperation mit unserem langjährigen Partner Agfa HealthCare haben wir verschiedene Deep Learning-Ansätze für die Analyse von Mammographie-Daten entwickelt, die den Workflow einer radiologischen Befundung auf verschiedenen Entscheidungsebenen unterstützen. Dafür wurden mehrere Deep Learning-Modelle kombiniert, die sich jeweils auf spezifische medizinische Fragestellungen fokussieren, wie beispielsweise die Detektion krankhafter Läsionen und Mikroverkalkungen oder ob eine Patientin dichteres Brustgewebe besitzt. Dadurch können wir insgesamt eine verbesserte Aussage auf Patientenebene treffen. Der aufgabenspezifische Aufbau der Lösung und die Möglichkeit auch auf Teilentscheidungen zuzugreifen, erhöht gleichzeitig die Erklärbarkeit und damit das Vertrauen in die Gesamtentscheidung. Um das Vertrauen in einzelne Black-Box Deep Learning-Komponenten unseres Modells und deren Entscheidungen zu stärken, wurden von uns weitere neue Methoden für die Erklärbarkeit von KI-Entscheidungen entwickelt.
Publikationen
Videos

Die exakte Segmentierung von Weichteiltumoren in multimodalen Daten ist in verschiedenen Bereichen der Medizin von großer Bedeutung, zum Beispiel für die Operations- und Biopsieplanung oder in der Radiotherapie. Hierfür werden multimodale Daten aufgenommen, wie MRI und PET/CT, wobei die Segmentierung der Tumore je nach Modalität und medizinischer Fragestellung unterschiedlich sein kann.
In Kooperation mit der Medizinischen Universität Wien haben wir verschiedene Methoden zur Segmentierung von Weichteiltumoren entwickelt und verglichen. Wir haben uns hierbei speziell auf die Modalitäts- und Fragestellungs-abhängige Tumorsegmentierung fokussiert, und wie wir mit der Fusion multimodaler Daten die Qualität der Segmentierung verbessern können.
Publikationen
Videos
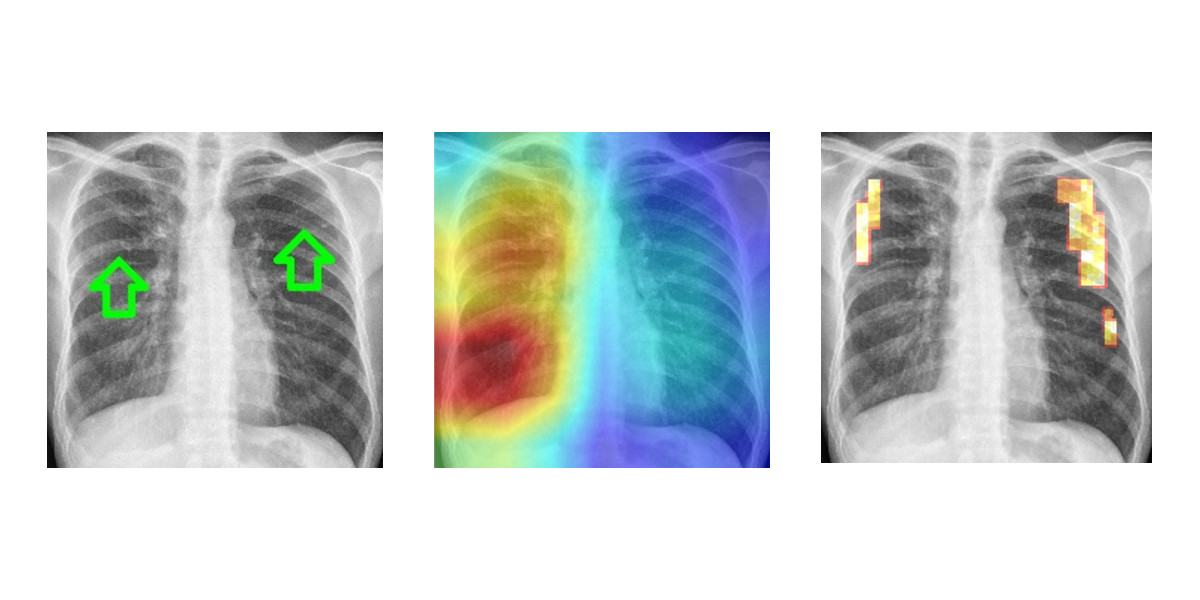
Um medizinisches Fachpersonal bei Patientenscreenings zur Erkennung von Tuberkuloseinfektionen zu unterstützen, hat VRVis gemeinsam mit Agfa Healthcare eine Deep Learning-basierte Anwendung entwickelt. Diese hilft gerade bei großen Patientenzahlen, erkrankte Personen schneller zu identifizieren, und so weitere Diagnose und Behandlungsabläufe zu optimieren. Weitere Informationen in der Pressemeldung.
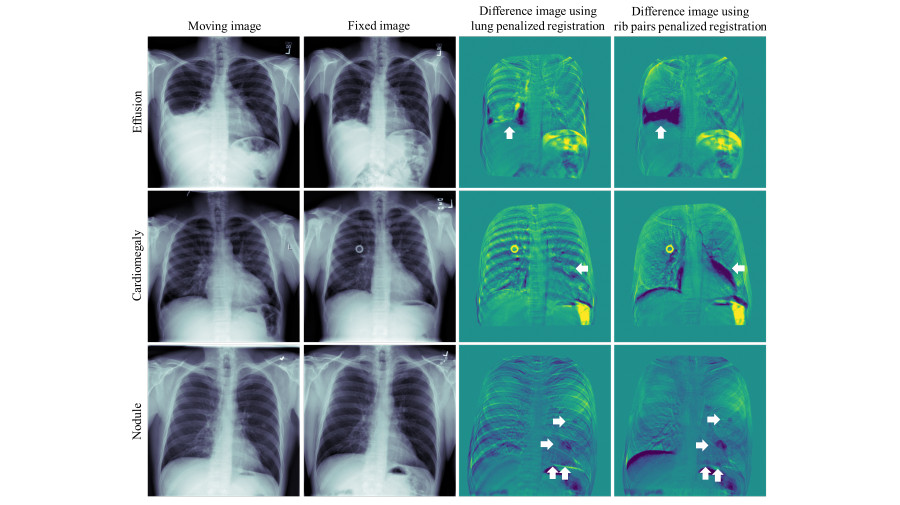
Die Registrierung von medizinischen Bildern ist ein wichtiger Bestandteil von medizinischen Analyse-Workflows. Sie ermöglicht es, verschiedene Bilder eines oder mehrerer Patienten direkt vergleichbar zu analysieren, indem die Bilder in dasselbe Koordinatensystem transferiert werden. Die Registrierung ist notwendig, um Bilder zwischen verschiedenen Zeitpunkten aufgenommenen Studien oder zwischen verschiedenen Modalitäten zu vergleichen.
Das VRVis entwickelte in Koorperation mit seinen Partnern aus der Medizin Methoden zur klassischen und Deep Learning-basierten Bildregistrierung und untersuchte insbesondere, wie Visualisierung Registrierungsworkflows unterstützen und Probleme aufzeigen kann.
Publikationen
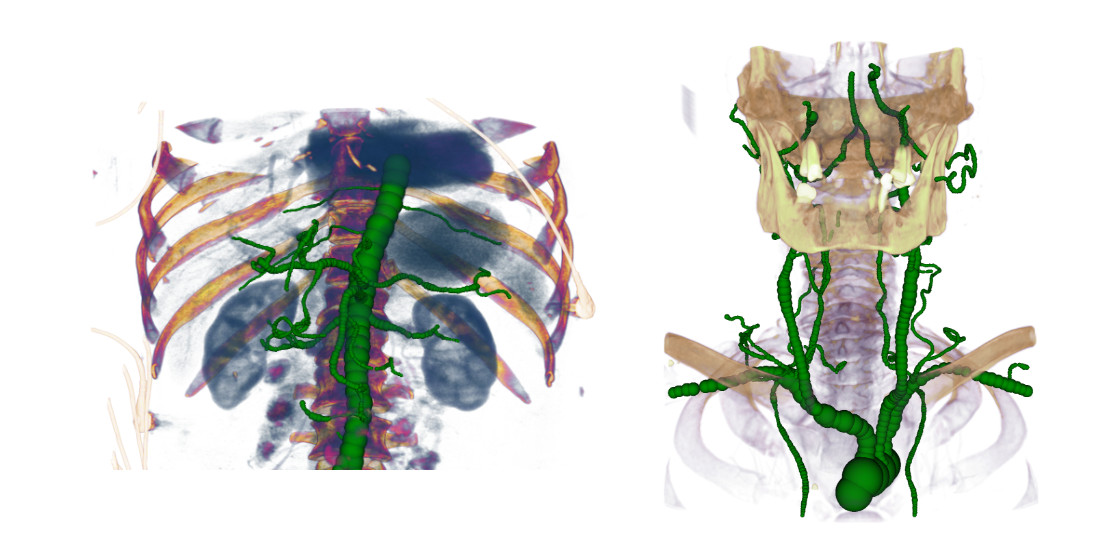
Arteriosklerose ist laut WHO weltweit die Todesursache Nummer 1. Eine detaillierte Analyse von Blutgefäßen in 3D-Angiographien sind im Allgemeinen sehr zeitaufwändig. VRVis hat gemeinsam mit AGFA Healthcare mehrere Methoden zur effizienten semi- und vollautomatischen Segmentierung von peripheren und koronaren Blutgefäßen, sowie deren semantischer Annotation, entwickelt, die teilweise sowohl prämiert als auch hochkarätig publiziert, patentiert und in Produkte unseres Kunden integriert wurden.
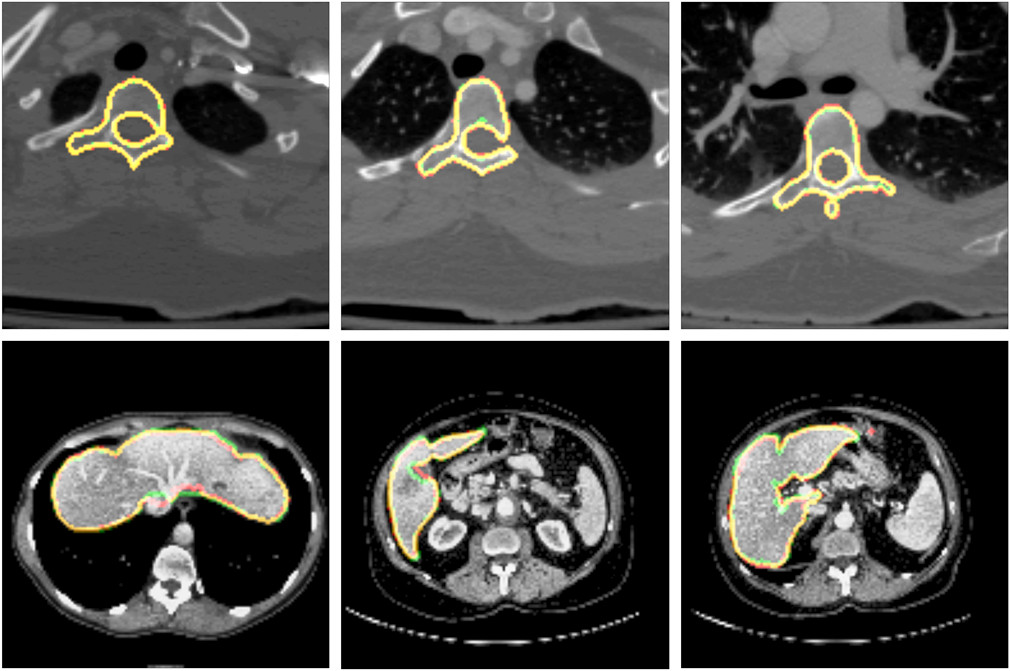
VRVis hat etliche grundlegende Methoden zur medizinischen Bildanalyse entwickelt und patentieren lassen. Darunter vielzitierte KI-Methoden zur gleichzeitigen Segmentierung mehrerer Objekte auf Röntgenbildern, sequenzielle Segmentierungsverfahren für CT-Bilddaten, effiziente Feature-Detektionsverfahren, sowie mehrere Grundlagenmethoden für statistische Form- und Texturmodelle, die z.B. auch heute noch in Kombination mit KI-Methoden für komplexe Aufgaben in der Organsegmentierung zur Anwendung kommen.
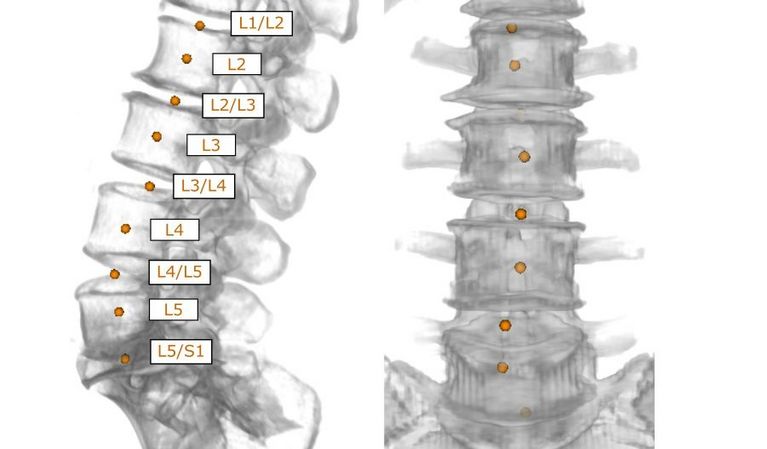
Die Wirbelsäule ist ein wichtiges Referenzsystem im menschlichen Körper, um bei einer radiologischen Untersuchung die Lage von Pathologien zu beschreiben. Dafür muss die anatomische Bezeichnung für jeden sichtbaren Wirbel im Bild bekannt sein. Diese muss in vielen Ländern von Radiologinnen und Radiologen manuell auf dem Bild eingetragen werden – eine zeitaufwändige Arbeit, die Radiologinnen und Radiologen zusätzlich zur eigentlichen Diagnose in einem knappen Zeitrahmen erledigen müssen. VRVis hat eine Gruppe von Algorithmen entwickelt, die menschliche Wirbel auf verschiedenen MR- und CT-Scans unabhängig vom Bildausschnitt vollautomatisch erkennen und anatomisch korrekt annotieren kann. Diese wichtige maschinelle Vorarbeit erleichtert Radiologinnen und Radiologen die Arbeit und verschafft mehr Zeit für die Diagnoseerstellung.

Herz-Kreislauf-Erkrankungen sind heute auch in Österreich die häufigste Todesursache. Eine detaillierte Analyse von (zeitabhängigen) 3D-Bilddaten des schlagenden Herzens und der Koronararterien eines Patienten ist extrem zeitaufwändig. Vollautomatische Methoden, die die Quantifizierung von Pathologien des Herzens erleichtern, können daher stark zur Beschleunigung der Befundung durch den Radiologen bzw. die Radiologin beitragen. VRVis hat bereits mehrere Lösungen für die Segmentierung der Herzkammern auf CT- und MR-Bildern sowie eine prämierte Lösung zur vollautomatischen Segmentierung von Koronararterien entwickelt.

Aus unserer hauseigenen Grundlagenforschung sind in Verbindung mit unserer anwendungsorientierten Forschungsarbeit für unseren Unternehmenspartner Agfa Healthcare viele Patente für KI-basierte Lösungen entstanden, beispielsweise zur vollautomatischen Segmentierung und Annotation von Blutgefäßen oder menschlichen Wirbeln. Erfahren Sie hier mehr über unsere Patente.
Die langfristige Vision dieses angewandten Forschungsprojekts ist die Nutzung der verfügbaren Datenressourcen zur Verbesserung der bildbasierten Diagnostik auf der Grundlage komplexer Daten in der täglichen klinischen Routine.
Das strategische Projekt bildet das Forschungsgruppen-übergreifende organisatorische und wissenschaftliche Zentrum für die Realisierung eines Integrative Visual Computing-Ansatzes.

VRVis ist Gründungsmitglied von Austrian BioImaging/CMI. Dies ist ein professionelles Konsortium aus verschiedenen österreichischen Forschungseinrichtungen und die offizielle Euro-BioImaging Initiative Österreichs.
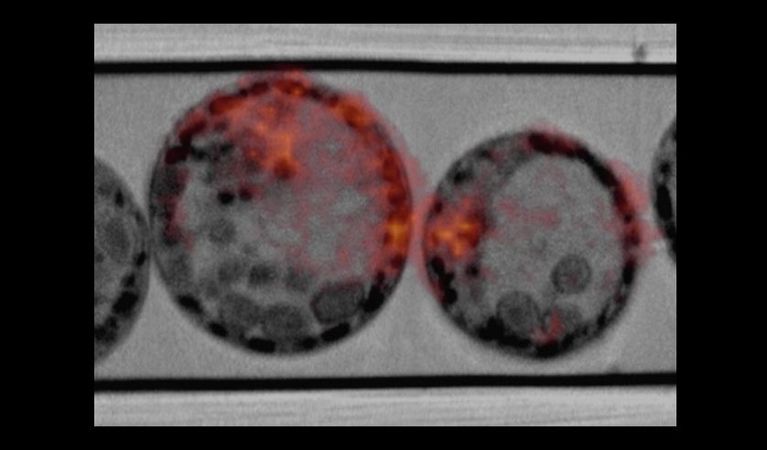
COMULIS (Correlated Multimodal Imaging in Life Sciences) ist eine EU-geförderte COST Action mit dem Ziel, Kollaborationen im Feld des Correlated Multimodal Imaging (CMI) voranzutreiben.
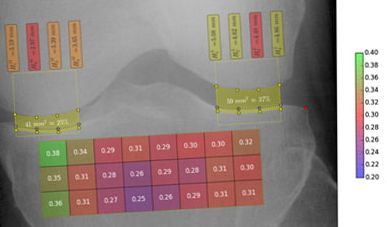
Visual Computing-Techniken zur automatisierten Erkennung von Osteoporose und Osteoarthritis.
Visual Computing für die Medizin: Bildverarbeitungslösungen für neue Anwendungen in der Radiologie.
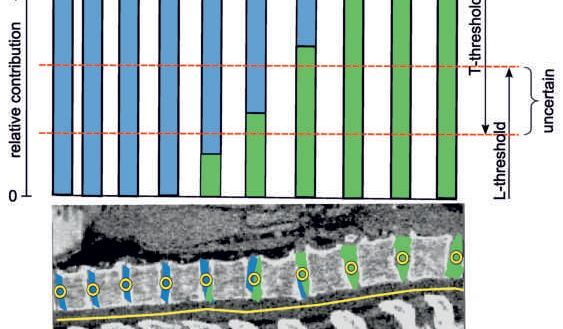
Die Analyse, Visualisierung und Erforschung von hochdimensionalen Bildräumen sind Gegenstand des KAFus-Projekts.

Workflows der nächsten Generation zur interaktiven Wissensgenerierung aus Bildern und Simulationen.

Software für die Verwendung von multimodalen Bildern in der externen Strahlentherapie.

Am 3. März 2020 wurde Katja Bühler, Leiterin unserer Biomedical Image Informatics-Gruppe, mit dem renommierten TU-Frauenpreis ausgezeichnet.

Visual Computing für die computergestützte Diagnostik und Operationsplanung.

Sebastian Zambal, Jiří Hladůvka, Armin Kanitsar, Katja Bühler, Shape and Appearance Models for Automatic Coronary Artery Tracking, WON MICCAI 2008 Contest: 3D Segmentation in Clinic: A Grand Challange.

C. Langer, M. Hadwiger, K. Bühler, Interaktive diffusionsbasierte Segmentierung von Volumendaten auf Grafikhardware, Bildverarbeitung für die Medizin 2005; GI Informatik Aktuell; Springer Verlag. pp 168-17, BVM 2005 Best Poster