
BrainWarp ist ein Tool, das die automatische Integration massiver Datensammlungen auf einen gemeinsamen Referenzraum – ein sogenanntes Standardgehirn – unterstützt, Funktionalitäten für die kollaborative Qualitätssicherung bereitstellt, sowie die nachfolgende Integration der Daten in BrainBase unterstützt.
BrainBase, BrainBaseWeb und BrainGazer ermöglichen aktuell die anatomische, räumliche oder strukturelle Suche auf sehr großen Sammlungen konfokaler Mikroskopiebilder von Gehirnen der Fruchtfiege, Fruchtfliegenlarve oder des Zebrafisches. Unserer patentierten Datenstrukturen erlauben eine intuitive Suche nach korrespondierenden Expressionsmustern von Neuronen auf zehntausenden von 3D Bildern, und das nahezu in Echtzeit. BrainGazer und BrainBaseWeb stellen zudem eine Vielzahl von Visual Analytics und Visualisierungstools für die Daten zur Verfügung.
Derzeit gibt es vier Instanzen der Software die öffentliche Datensammlungen unterstützen:
www.larvalbrain.org: Standard Version des Larvalen Fruchtfiegengehirns (drosophila melanogaster) mit anatomischen Annotationen, sowie einer Sammlung genetischer Linien. Die Datensammlung wurden gemeinsam von der Universität Leipzig (Prof. Andreas Thum), der UCLA (Prof Hartenstein), der RWTH Aachen (Prof. Dorit Merhof) und dem VRVis (Dr. Katja Bühler) im Rahmen eines von der DFG und dem FWF geförderten Projektes entwickelt und bereitgestellt.
fruitfly.tefor.net und zebrafish.tefor.net: Sammlung konfokaler Mikroskopiebilder mit neuralen Strukturen, die von der TEFOR Infrastruktur bereitgestellt werden.
https://implegacy.brainbase.at: Ältere Version von BrainBaseWeb, die eine Sammlung von über 15.000 3D Bildern der VT Linien in der adulten Fruchtfliege (drosophila melanogaster) zeigen, die von Barry Dickson am IMP in Wien generiert wurden.
BrainTrawler – integratives Analyse-System für komplexe Gehirnnetzwerke:
Zusammen mit Neurowissenschaftlerinnen und -wissenschaftlern des Haubensak Labs des IMP Wien und Boehringer Ingelheim entwickeln wir BrainTrawler, ein webbasierendes Framework zur Untersuchung heterogener neurobiologischer Daten von Mäusen und Menschen. Mittels räumlicher Indexierung ermöglichen wir die Analyse von Genexpressiondaten und komplexen Gehirnnetzwerken in Echtzeit. Eine Zuordnung der Daten zu hierarchisch organisierten anatomischen Strukturen ermöglicht die Suche auf verschiedenen anatomischen Ebenen. Zusammen mit intuitiver Netzwerk-Visualisierung, iterativen visuellen Abfragen und quantitativen Informationen ermöglicht BrainTrawler die genetische Dissektion multimodaler Netzwerke auf lokaler/globaler Ebene im räumlichen Kontext. Weitere Infos hier.
Lizenzinformationen sowie Informationen über die Zusammenarbeit an einem gemeinsamen Forschungsprojekt sind auf Anfrage erhältlich. Bitte kontaktieren Sie DI Dr. Gerd Hesina unter products(at)vrvis.at!
Mehr über die Forschungsarbeit unserer Biomedical Image Informatics-Forschungsgruppe.

Am 3. März 2020 wurde Katja Bühler, Leiterin unserer Biomedical Image Informatics-Gruppe, mit dem renommierten TU-Frauenpreis ausgezeichnet.

Im Rahmen der 40-Jahres-Feier der Österreichischen Computer Gesellschaft (OCG) am 9. Juni 2015 wurde Johannes Sorger mit dem diesjährigen OCG-Förderpreis ausgezeichnet.

Das Paper "Visual and Quantitative Analysis of Higher Order Arborization Overlaps for Neural Circuit Research" wurde ausgezeichnet.

Das webbasierte Tool "neuroMAP" des VRVis und IMP erhielt die Best Paper-Auszeichnung der IEEE BioVis 2013.

Im Projekt Larvalbrain 2.0 soll durch die Integration von Daten über strukturelle, molekulare, physiologische und verhaltensbedingte Ergebnisse von Drosophila-Melanogaster-Larven ein dynamischer, mehrstufiger Gehirnatlas etabliert werden.
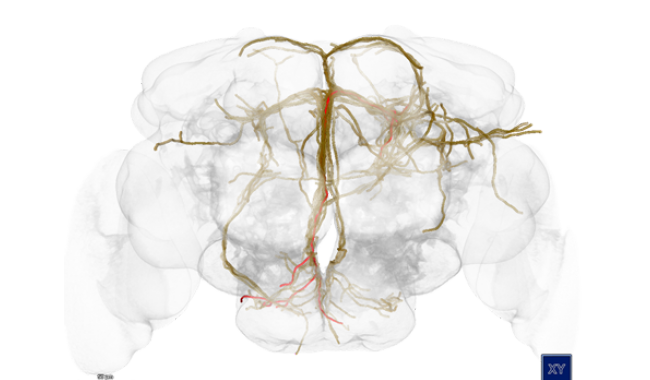
Zu verstehen, wie das Gehirn funktioniert, ist eine der größten Herausforderungen, mit denen sich die Neurowissenschaften heute befassen. Dazu bedarf es spezieller Software-Infrastrukturen, um die Erforschung des komplexen Zusammenspiels von Genen, Struktur und Funktion zu ermöglichen und zu beschleunigen.

VRVis steuert Datenanalyse- und Visualisierungstools bei, die auf die Unterstützung und Beschleunigung der Forschung des Instituts für Molekulare Pathologie Wien zugeschnitten sind.

Seit Jahrhunderten kartografieren die Neurowissenschaften das Gehirn. Bisher erwies sich der Schritt von einfachen Karten zu einem allgemein akzeptierten Modell als äußerst schwierig. In diesem Projekt wird ein 4D-Atlas des Gehirns der Fruchtfliegenlarve gebaut.
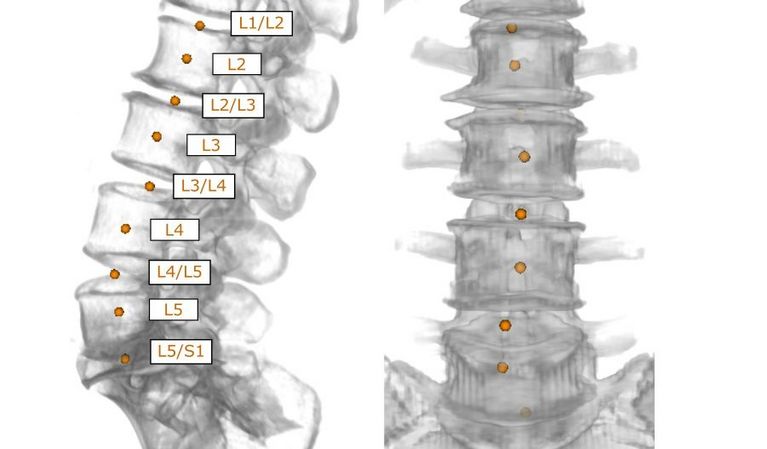
Die langfristige Vision dieses angewandten Forschungsprojekts ist die Nutzung der verfügbaren Datenressourcen zur Verbesserung der bildbasierten Diagnostik auf der Grundlage komplexer Daten in der täglichen klinischen Routine.
Das strategische Projekt bildet das Forschungsgruppen-übergreifende organisatorische und wissenschaftliche Zentrum für die Realisierung eines Integrative Visual Computing-Ansatzes.