

Seit 2008 entwickelt VRVis komplette Plattformen zum webbasierten Management, der Visualisierung, der Integration und des Minings von Gehirndaten. Alleinstellungsmerkmal des Systems sind die sehr performanten räumlichen Indexingmethoden, die es intuitiv und fast in Echtzeit erlauben, den Inhalt zehntausender von 3D Datensätzen nach ähnlichen oder überlappenden Sub-Strukturen zu durchsuchen. Unsere Entwicklungen basieren auf unserem modularen Brain* Framework.
Unsere Systeme sind mittlerweile in etlichen Institutionen installiert und bieten teilweise auch öffentlichen Zugang zu großen Datensammlungen. Das BrainBase System hosted zur Zeit Datensammlungen zum larvalen und adulten Gehirn der Drosophila melanogaster und des Zebrafischs. (siehe z.B. larvalbrain.org, fruitfly.tefor.net, zebrafish.tefor.net)
Zusammen mit Neurowissenschaftlerinnen und -wissenschaftlern des Haubensak Labs und Boehringer-Ingelheim entwickeln wir BrainTrawler für Datensammlungen zum Gehirn von Mäusen und Menschen. Mittels räumlicher Indexierung ermöglichen wir hier die Analyse von Genexpressiondaten und komplexen Gehirnnetzwerken in Echtzeit. Eine Zuordnung der Daten zu hierarchisch organisierten anatomischen Strukturen ermöglicht die Suche auf verschiedenen anatomischen Ebenen. Zusammen mit intuitiver Netzwerk-Visualisierung, iterativen visuellen Abfragen und quantitativen Informationen ermöglicht BrainTrawler die genetische Dissektion multimodaler Netzwerke auf lokaler/globaler Ebene im räumlichen Kontext.
Auswahl an Publikationen
Projekt: 4D Larvalbrain
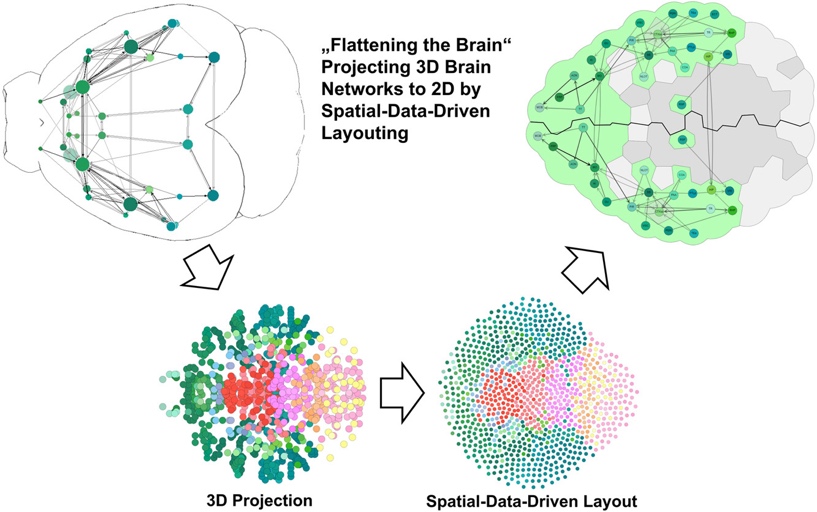
Die Visualisierung neurowissenschaftlicher Daten für Publikationen, aber auch für die interaktive Datenexploration im Kontext des Brain*-Systems ist ein wichtiger Forschungsbereich des VRVis. In mehreren Arbeiten wurde beispielsweise ein besonderer Schwerpunkt auf die Visualisierung und die interaktive Analyse von Gehirnnetzwerkdaten gelegt, sowie auf die Umsetzung sehr performanter webbasierter 2D- und 3D- Visualisierung von Gehirndaten (siehe z.B. larvalbrain.org).
Wir entwickeln und erforschen maßgeschneiderte Data Science und Visual Analytics-Lösungen, um die oft sehr anspruchsvollen und heterogenen Daten aus Verhaltens- und pharmakologischen Experimenten zu analysieren. Die Datentypen sind vielfältig: Neben Aufzeichnungen neuronaler Aktivitäten wenden wir unsere Methoden auch auf Daten an, die aus Videoaufnahmen von Verhaltensstudien extrahiert wurden.
Neben "klassischen" Methoden des Machine und Deep Learning, wie Klassifizierungen und Clusteranalysen, verwenden wir auch Methoden aus der Informationstheorie oder psychophysikalische Modelle.
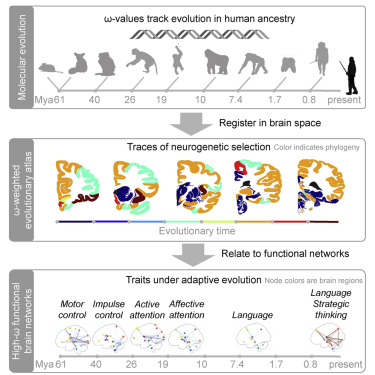
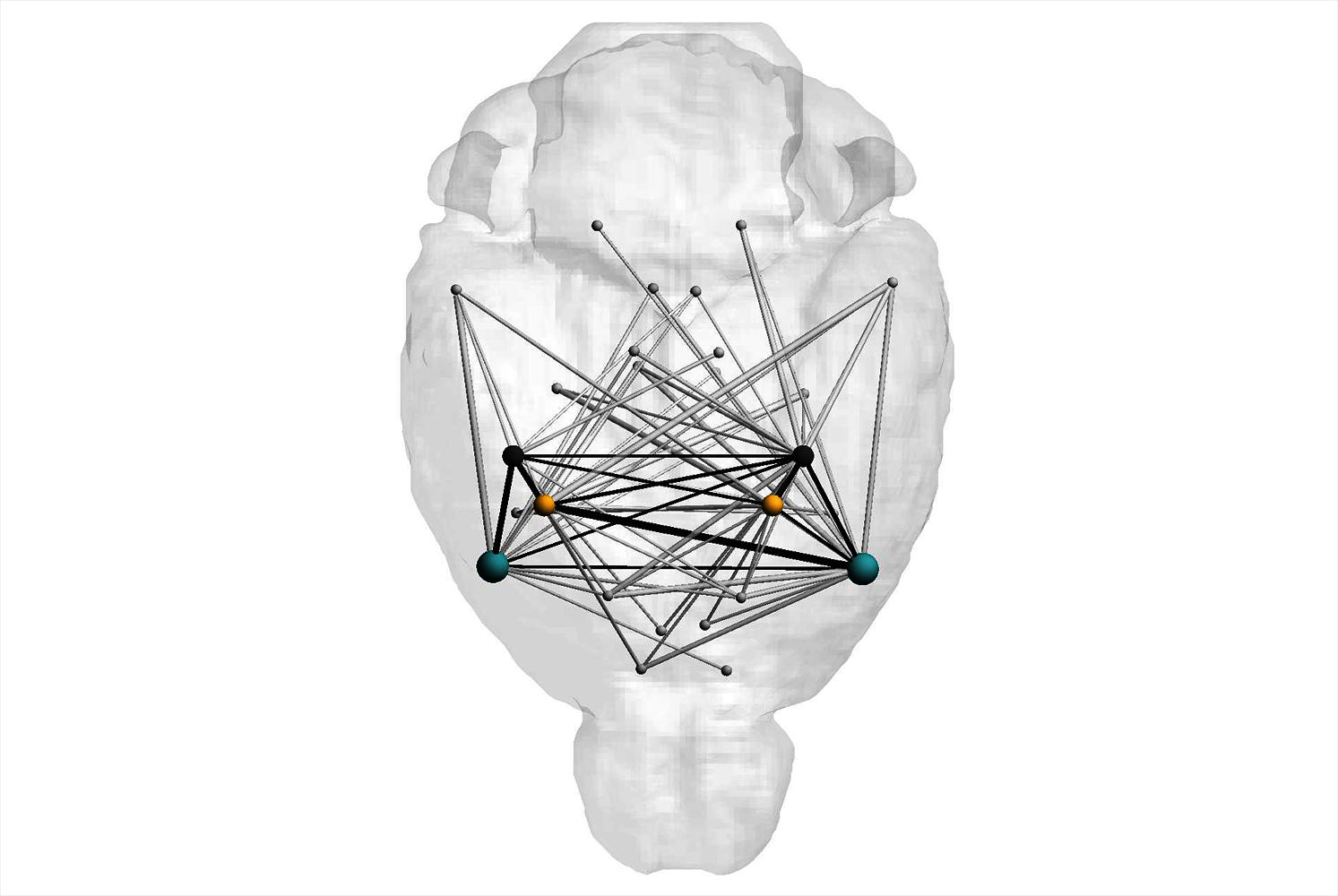
Computational Neuroscience verwendet ausschließlich Daten und mathematische Methoden bzw. Methoden der Informatik, um neue Erkenntnisse zu gewinnen. Gemeinsam mit mehreren Partnerinnen und Partnern haben wir beispielsweise neue Ansätze entwickelt, um neue funktionale Gehirnregionen auf der Basis zehntausender Bilddatensätze von Neuronen zu berechnen oder funktionale Teilnetzwerke, die mit bestimmten Genen korrelieren, zu rekonstruieren. In einer aktuellen Arbeit versuchen wir den Ursprung menschlicher kognitiver Merkmale zu kartieren, indem wir evolutionäre Genomik mit funktioneller Neuroanatomie kombinieren, um die neurogenetische Entwicklung der menschlichen Hirnfunktionen direkter und umfassender zu rekonstruieren.
Um eine akzeptable Performance für die komplizierten Berechnungen auf sehr großen Datenmengen zu erreichen, entwickeln wir hochparallelisierte Lösungen und nutzen gezielt verfügbare High Performance Computing-Infrastrukturen.
Referenzen
Extended Reality-Werkzeuge (Augmented und Virtual Reality) finden immer öfter Einsatz in den Life Sciences. Denn digitale Realitäten helfen wissenschaftliche Experimente, Trainings und Behandlungen zu verbessern, indem sie immersive und realitätsgetreue Umgebungen schaffen, die kostengünstig und kontrollierbar sind.
Für die Abteilung für Neurobiologie der Universität Wien entwickelten wir am VRVis einen Bewegungs- und Kamera-Aufbau, mit welchem die Reaktionen von Jagdspinnen untersucht werden können. Die Spinne läuft auf einer Kugel von ein Meter Durchmesser in einer projizierten Umgebung. Eine Kamera misst die Bewegungen der Spinnen und dreht die Kugel um die Spinne im Zentrum zu halten. Dadurch können beliebige visuelle Stimuli in Abhängigkeit der Spinnenbewegung erzeugt werden, auch solche, die mit einem physischen Aufbau unmöglich wären.
Referenzen

Im Projekt Larvalbrain 2.0 soll durch die Integration von Daten über strukturelle, molekulare, physiologische und verhaltensbedingte Ergebnisse von Drosophila-Melanogaster-Larven ein dynamischer, mehrstufiger Gehirnatlas etabliert werden.

Seit Jahrhunderten kartografieren die Neurowissenschaften das Gehirn. Bisher erwies sich der Schritt von einfachen Karten zu einem allgemein akzeptierten Modell als äußerst schwierig. In diesem Projekt wird ein 4D-Atlas des Gehirns der Fruchtfliegenlarve gebaut.
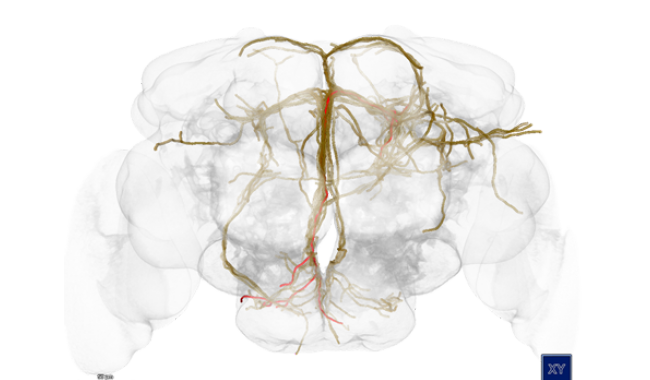
Zu verstehen, wie das Gehirn funktioniert, ist eine der größten Herausforderungen, mit denen sich die Neurowissenschaften heute befassen. Dazu bedarf es spezieller Software-Infrastrukturen, um die Erforschung des komplexen Zusammenspiels von Genen, Struktur und Funktion zu ermöglichen und zu beschleunigen.

VRVis steuert Datenanalyse- und Visualisierungstools bei, die auf die Unterstützung und Beschleunigung der Forschung des Instituts für Molekulare Pathologie Wien zugeschnitten sind.
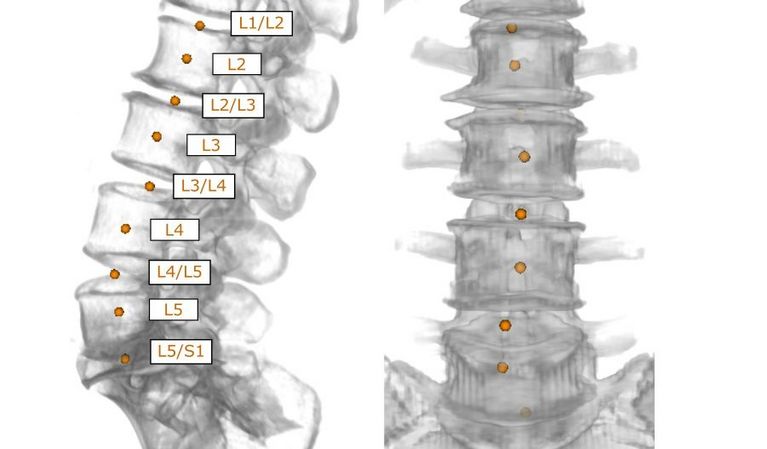
Die langfristige Vision dieses angewandten Forschungsprojekts ist die Nutzung der verfügbaren Datenressourcen zur Verbesserung der bildbasierten Diagnostik auf der Grundlage komplexer Daten in der täglichen klinischen Routine.
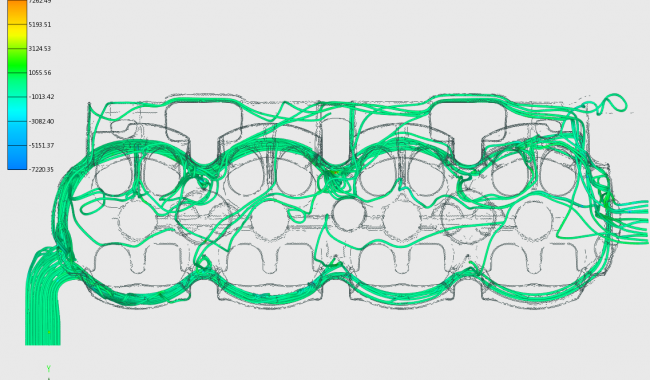
Das strategische Projekt bildet das Forschungsgruppen-übergreifende organisatorische und wissenschaftliche Zentrum für die Realisierung eines Integrative Visual Computing-Ansatzes.
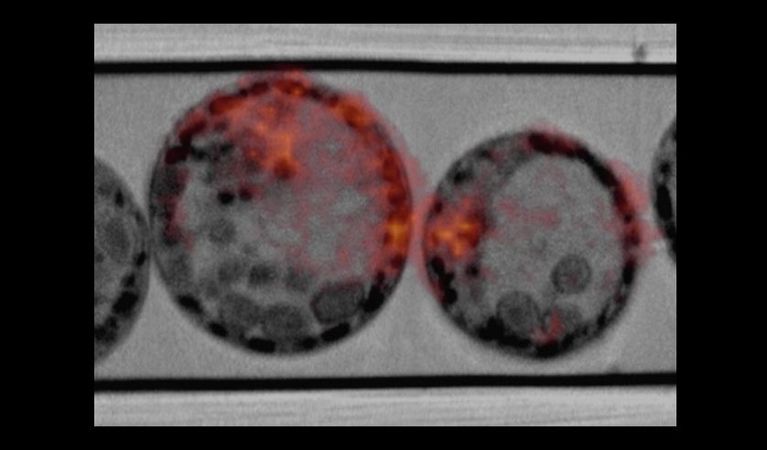
COMULIS (Correlated Multimodal Imaging in Life Sciences) ist eine EU-geförderte COST Action mit dem Ziel, Kollaborationen im Feld des Correlated Multimodal Imaging (CMI) voranzutreiben.

VRVis ist Gründungsmitglied von Austrian BioImaging/CMI. Dies ist ein professionelles Konsortium aus verschiedenen österreichischen Forschungseinrichtungen und die offizielle Euro-BioImaging Initiative Österreichs.

Im Rahmen der 40-Jahres-Feier der Österreichischen Computer Gesellschaft (OCG) am 9. Juni 2015 wurde Johannes Sorger mit dem diesjährigen OCG-Förderpreis ausgezeichnet.

Das Paper "Visual and Quantitative Analysis of Higher Order Arborization Overlaps for Neural Circuit Research" wurde ausgezeichnet.

Das webbasierte Tool "neuroMAP" des VRVis und IMP erhielt die Best Paper-Auszeichnung der IEEE BioVis 2013.